リボソームの"腕"様分子 stalkタンパク質の研究 (内海)
※内海先生は平成31年3月31日をもって退職いたしました。
真核細胞ではリボソームのもとで、1秒間に数個のアミノ酸がつながりタンパク質が作られています。材料となるアミノ酸は一つ一つ特定のtRNAに結合した状態でリボソームの機能部位(Aサイト)に次々に運ばれペプチド鎖が伸長していきます。一連のプロセスには複数の補助因子(GTPase翻訳因子)がリボソーム上の1カ所の結合部位と交互に相互作用する必要があり、この相互作用の効率的切り換えがタンパク質合成速度と密接に関係します。
リボソームと翻訳因子間の相互作用にリボソーム中に複数コピーが存在する柔軟なストークタンパク質が寄与することは1970年代より知られていましたがその作用機構については未解決のまま残されていました。
当研究グループは、真核生物ストークタンパク質と構造面で類似する古細菌のサンプルを用い、古細菌aP1ストークはホモダイマーを形成し、その3個がaP0と呼ばれるタンパク質と結合し、ユニークな7量体を形成することを報告してきました。
そして当研究グループは、北海道大学の田中勲・姚閔研究室との共同研究により以下のことを明らかにしました。
1.各aP1ダイマーが、aEF-2、aEF-1aおよびaIF5BのGTPase翻訳因子群と直接結合すること
2.その結合にリボソーム表面で広範囲に運動しているaP1のC末端部位が寄与すること
3.結合にC末端部位の4個の疎水性アミノ酸を介すること
(Nomura, N. et al. Archaeal ribosomal stalk protein interacts with translation
factors in a nucleotide-independent manner via its conserved c terminus.
Proceedings of the National Academy of Sciences of the United States of
America (2012). URL http://dx.doi.org/10.1073/pnas.1112934109.)
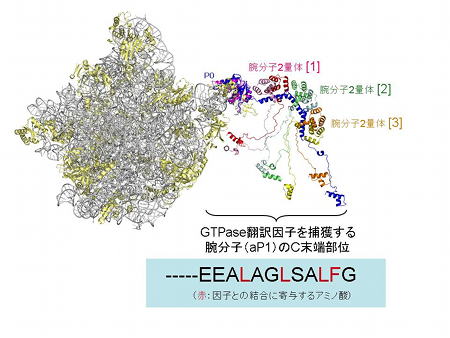
リボソーム腕様タンパク質(ストークタンパク質)複合体のモデル
古細菌50Sサブユニットの結晶構造 (左;Klein et al. 2001) に腕様タンパク質複合体の構造(右;Naganuma et al.
2010)を適合させたモデル(リボソームには長い腕様構造が含まれることがわかる)。aP1のC末端部分の構造はアミノ酸配列(下)から推測したモデル。翻訳因子との結合に重要と判明したアミノ酸を赤で示す(下)。
遺伝情報発現に関わる生体分子の立体構造解析 (伊東) 新HP
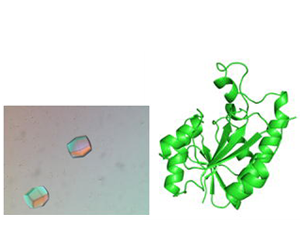
DNA上の遺伝情報が、生命活動の実際の働き手であるタンパク質やRNAへと変換される反応(遺伝情報の発現)の仕組みを解き明かすことは、生命科学の中心的なテーマの1つです。我々は、遺伝情報の発現に関わる生体分子の立体構造をX線結晶構造解析により決定し、生化学的解析と併せて、それらの反応のメカニズムを原子分解能レベルで解明することを目指しています。
主な研究テーマ
- ペプチジルtRNA加水分解酵素の構造・機能解析
- リボソームストークタンパク質の構造・機能解析
- 小分子RNA関連酵素の構造・機能解析